基因组测序/芯片
基因组测序/芯片 1. 背景介绍
外显子(exon)是真核生物基因的一部分,包含着合成蛋白质所需要的信息。全部外显子被称为“外显子组”(Exome)。外显子组测序(Exome sequencing)是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。由于外显子组测序捕获目标区域只占人类基因组长度的约1%,因此远比进行全基因组序列测序来得更简便、经济,目标区域覆盖度也更高,便于变异检测。
该项技术可用于以下研究
1)检测疾病样本中外显子区域内高风险碱基变异位点;
2)配合大样本分析,确定孟德尔遗传疾病相关外显子SNP位点和基因;
3)在癌症研究过程中,检测癌症样本外显子区域内的体细胞突变位点和潜在的融合基因;
4)用于种群遗传学研究的大规模样本基因组分析,检测SNP位点、LD并绘制种群图谱。
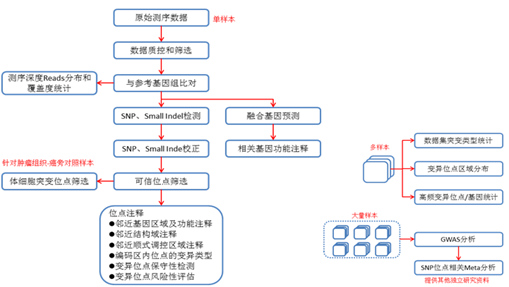
标准分析:
1) 数据质控和筛选;
2) 与参考基因组比对,得到测序深度Reads分布和覆盖度统计;
3) SNP、small Indel检测
4) SNP、small Indel矫正
高级分析:
1) 可信位点筛选;
2) 体细胞突变位点筛选(癌症)
3) 位点注释(邻近基因区域与功能注释、邻近结构域注释、邻近顺势调控区域注释、编码区内位点的变异类型;变异位点保守性检测、变异位点风险评估)
4) 数据集图片类型统计(多样本);
5) 变异位点区域分布(多样本);
6) 高频变异位点/基因统计(多样本);
7) GWAS分析(大样本量);
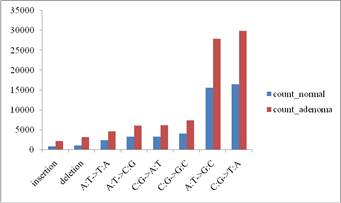
示例图1 各类型SNV在样本中的个数统计
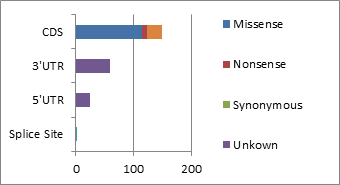
示例图2 不同类型外显子区域上的SNV类型统计
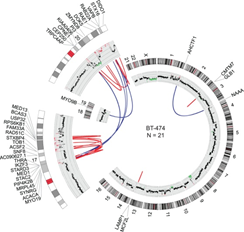
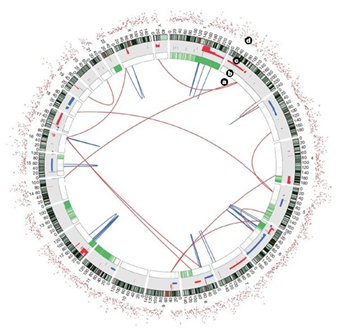
示例图3 融合基因预测
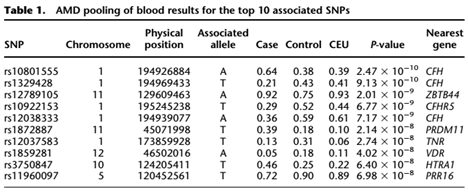
示例图 大量样本的GWAS分析结果
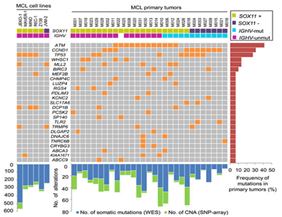
示例图 肿瘤样本高频率突变基因统计

全基因组重测序是基于Illumina测序平台,对已有的参考基因组序列的物种进行个体或群体的全基因组测序,结合高通量测序和生物信息学分析方法,识别、发现de novo的somatic和germ line突变,包括单个核苷酸多态性位点(SNP)、插入缺失(InDel)突变、结构变异(SNV)、拷贝数变异(CNV)、杂合性缺失(LOH)、融合基因发现等多态性信息,获得生物学遗传特征,进行遗传进化分析,预测与进化选择相关的候选基因mutation之间的关系。
收集样品和采集信息后,提取样本基因组DNA(或者客户提供提取好的基因组DNA),对提取的基因组DNA进行质量检测(检测项目包括抽提的DNA的纯度、浓度等质量问题)。对检验合格的样品进行文库制备和检测。对质检合格的文库进行上机测序,流程图如图所示:
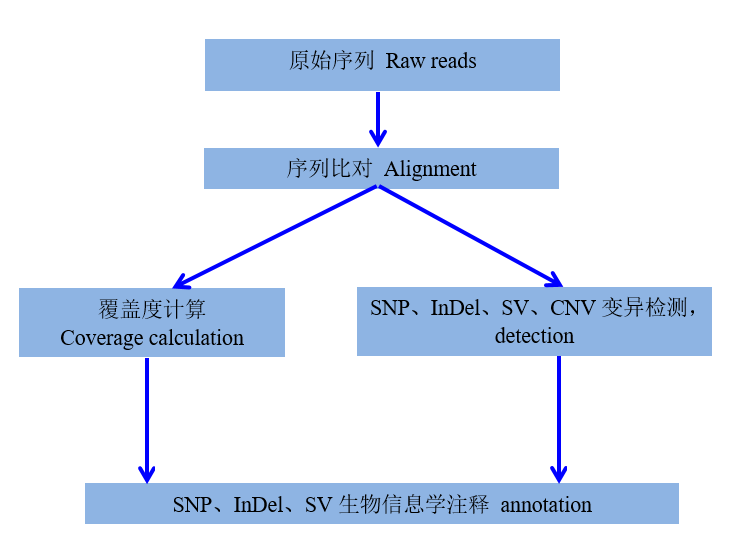
1) 样本准备
样品要求:DNA的OD值(260/280)在1.8-2.0之间,无RNA污染;
DNA浓度≥50 ng/μL,每个样品的DNA总量≥15μg;
DNA样品需溶解在H2O或者TE(pH8.0)中,低温、密封运输(-20℃,或者干冰)
2) 测序深度
(1)SNP变异检测——5X以上;
(2)鉴定绝大部分SNPs——30X;
(3)SV检测——10X以上;
(4)癌症组织中较大的SV——50X以上;
(5)群体重测序——10X;
3) 分析内容
(1) 测序数据统计及质量评估;
(2) SNP检测及在基因组中的分布;
(3)InDel检测及在基因组的分布;
(4)Structure Variation检测及在基因组中的分布;
(5)移码突变的分布;
(6)融合基因的发现;
(7)DNA水平差异基因分析;
(8)遗传进化分析(多样本);
(9)单体型预测(多样本);
(10)全基因组关联分析(多样本);
(1)SNP突变趋势与模式分析
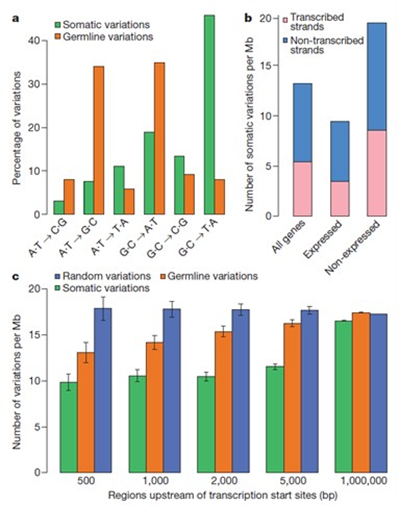
(2)SNP在染色体上的定位分析
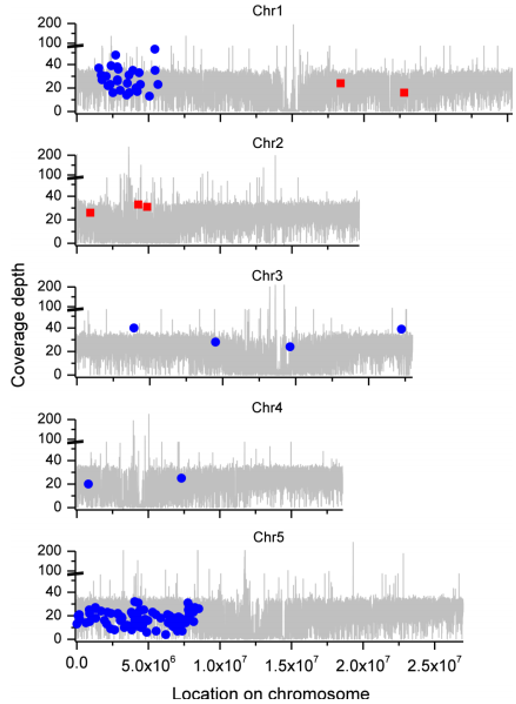
(3)SV偏好性分析
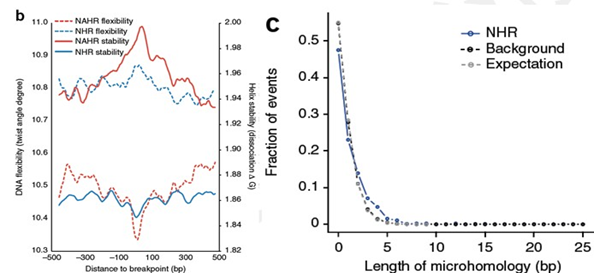
(4)CNV在染色体上分布
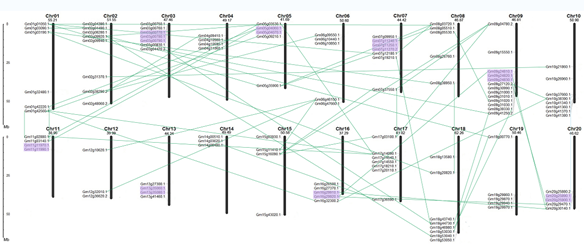
(5)全基因组全局重排分布特征分析